HAPLOOTYPE STUDY METODER, DIAGNOSTICS, Sjukdomar
- 1719
- 264
- Erik Eriksson
En Haplootype Det är en region i genomet som tenderar att ärva tillsammans genom flera generationer; Vanligtvis finns allt i samma kromosom. Haplootyper är produkten av genetisk bindning och förblir intakt under genetisk rekombination.
Ordet "haplotipo" härrör från en kombination av ordet "haploid" och ordet "genotyp". "Haploid" hänvisar till celler med en enda uppsättning kromosomer och "genotyp" talar om den genetiska sammansättningen av en organisme.
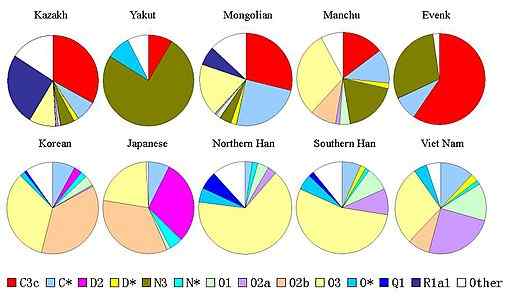
 Schema för distributionen av kromosom haplotyper och i asiatiska populationer (källa: Moogalord [Public Domain] via Wikimedia Commons) efter definitionen kan en haplotyp beskriva ett par gener eller mer som ärvts tillsammans på en kromosom från en föräldra, eller kan beskriva en kromosom som är helt ärft från en föräldraskap, som är fallet med kromosomen och män.
Schema för distributionen av kromosom haplotyper och i asiatiska populationer (källa: Moogalord [Public Domain] via Wikimedia Commons) efter definitionen kan en haplotyp beskriva ett par gener eller mer som ärvts tillsammans på en kromosom från en föräldra, eller kan beskriva en kromosom som är helt ärft från en föräldraskap, som är fallet med kromosomen och män.
Till exempel, när haplotyper delar gener för två olika fenotypiska karaktärer, såsom hårfärg och ögonfärg, kommer individer som har hårfärggen också att ha den andra genen för ögonens färg.
Haplootyper är ett av de mest använda verktygen idag för att studera släktforskning, för att spåra sjukdomens ursprung, för att karakterisera den genetiska variationen och fylogeografin för populationer av olika typer av levande varelser.
Det finns flera verktyg för att studera halootyper, ett av de mest använda idag är "Haplotypkarta”(Hapmap), som är en webbsida som gör det möjligt att bestämma vad som är genomsegmenten som är haplotyper.
[TOC]
Studiemetoder
Haplootyper representerar en möjlighet att förstå arvet av gener och deras polymorfism. Med upptäckten av tekniken "Polymeras -kedjereaktion" (PCR, engelska "Polymeraskedjereaktion”) Han avancerade allmänt i studien av haplotyper.
Det finns för närvarande många metoder för att studera haplotyper, några av de mest höjdpunkterna är:
DNA -sekvensering och enstaka nukleotidpolymorfismdetektering (SNP)
Utvecklingen av ny generation av sekvenseringsteknologier representerade ett stort språng för studier av haplotyper. Ny teknik tillåter variationer av upp till en enda nukleotidbas i specifika regioner i en haplotyp.
Kan tjäna dig: Rekombinant DNA: Teknik, applikationer och stiftelserI bioinformatik används termen haplotyp också för att hänvisa till arvet av en enda nukleotidpolymorfismer (SNP) i DNA -sekvenser.
Att kombinera bioinformatikprogram med upptäckt av haplotyper med hjälp av ny generationssekvensering kan exakt identifieras positionen, ersättningen och effekten av förändring av varje bas på genomens genom genom en befolkning.
Microsatellites (SSR)
Microsatelliterna eller SSR: er härstammar från det engelskaimplementera upprepning och Kort tandemupprepning". Dessa är korta nukleotidsekvenser som upprepas successivt inom ett genomregion.
Det är vanligt att hitta mikrosatelliter i de icke -kodande haplotyper, därför kan olika alleler genom att upptäcka variationer i antalet repetitioner av mikrosatelliter observeras i individuella haplotyper.
Mikrosatellitmolekylära markörer har utvecklats för att upptäcka oändliga haplotyper, från kön av växter som papaya (Karikapaya) tills upptäckten av mänskliga sjukdomar som falciform anemi.
Polymorfismer av de amplifierade fragmenten (AFLP)
Denna teknik kombinerar amplifiering med PCR -reaktioner med DNA -matsmältning med två olika restriktionsenzymer. Tekniken upptäcker polymorfa loci i haplootyper enligt de olika skärplatserna i DNA -sekvensen.
För att bättre illustrera tekniken, låt oss föreställa oss tre tygfragment av samma längd, men skärs till olika platser (dessa fragment representerar tre fragment av förstärkta haplotyper med PCR -tekniken).
När tyget skärs kommer många delar av olika storlek att erhållas, eftersom varje trasa skärs på olika platser. När vi beställer fragmenten enligt vilken typ av tyg som de kommer, kan vi observera var skillnaderna mellan tyger eller haplotyper finns.
Diagnostik och sjukdomar
En viktig fördel med den genetiska studien av haplotyper är att dessa förblir nästan intakta eller oförändrade i tusentals generationer, och detta möjliggör identifiering av avlägsna förfäder och var och en av de mutationer som individer bidrar till utvecklingen av sjukdomar.
Kan tjäna dig: fenotypiska variationerHaplootyper i mänskligheten varierar beroende på raser och från denna skopa har gener upptäckts inom haplotyper som orsakar allvarliga sjukdomar i var och en av de mänskliga raserna.
I projektet Hapmap Fyra rasgrupper ingår: europeiska, nigerianska yoruba, kinesiska Han och japanska.
På detta sätt projektet Hapmap Det kan täcka de olika grupperna av populationer och spåra ursprunget och utvecklingen av många av de ärftliga sjukdomarna som påverkar var och en av de fyra raserna.
En av de sjukdomar som oftast diagnostiseras med hjälp av haplotypanalys är falciform anemi hos människor. Denna sjukdom diagnostiseras genom att spåra frekvensen av afrikanska haplotyper till en befolkning.
Att vara en sjukdom som ursprungligen är från Afrika, och identifierar afrikanska haplotyper i populationer gör att du enkelt kan spåra människor som har mutationen i den genetiska sekvensen för beta -globinas av erytrocyter i form av en HOZ (kännetecken för patologi).
Exempel
Med haplotyper fylogenetiska träd som representerar evolutionära förhållanden mellan var och en av de haplotyperna som finns i ett prov av homologa DNA -molekyler eller samma art, i en region som har liten eller ingen rekombination konstrueras.
En av de mest studerade grenarna genom haplotyper är utvecklingen av människors immunsystem. HAPLOOTYPER SOM KODE FÖR TOLL -TYPE -receptorn (en nyckelkomponent i det medfödda immunsystemet) har identifierats för Neanderthal och Denisovanos genomet.
Detta gör att de kan spåra hur genetiska sekvenser har förändrats i populationerna av "moderna" människor med avseende på haplotypsekvenserna som motsvarar "förfäder" människor.
Bygga ett nätverk av genetiska relationer från mitokondriell haplotyp.
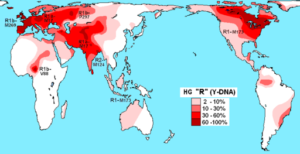 HAPLOGROUP DISTRIBUTION R (DNA-Y) i infödda populationer (Källa: Maucioni [CC av 3.0 (https: // CreativeCommons.Org/licenser/av/3.0)], via Wikimedia Commons) Mångfalden av haplotyper används för att spåra och studera den genetiska mångfalden hos djur som är uppvuxna i fångenskap. Dessa tekniker används speciellt för svåra arter för att övervaka vilda djur.
HAPLOGROUP DISTRIBUTION R (DNA-Y) i infödda populationer (Källa: Maucioni [CC av 3.0 (https: // CreativeCommons.Org/licenser/av/3.0)], via Wikimedia Commons) Mångfalden av haplotyper används för att spåra och studera den genetiska mångfalden hos djur som är uppvuxna i fångenskap. Dessa tekniker används speciellt för svåra arter för att övervaka vilda djur.
Djurarter som hajar, fåglar och stora däggdjur som jaguarer, elefanter, bland annat utvärderas ständigt genetiskt genom mitokondriella haplotyper för att övervaka populationens genetiska tillstånd i fångenskap.
Referenser
- Bahlo, m., Stankovich, J., Hastighet, t. P., Rubio, j. P., Burfoot, r. K., & Fot, s. J. (2006). Deteterande genom bred haplotypdelning med SNP eller Microsatellite Haplotype -data. Human Genetics, 119 (1-2), 38-50.
- Dannemann, m., Andrés, a. M., & Kelso, J. (2016). Introgress av neandertal-ando Denisovan-liknande haplotyper bidrar till adaptiv variation i mänskliga avgiftsliknande mottagare. American Journal of Human Genetics, 98 (1), 22-33.
- Från Vries, h. G., Van der meulen, m. TILL., Rozen, r., Halley, D. J., Scheffer, h., Leo, s.,... & du Meerman, G. J. (nitton nittiosex). Haplotypidentitet mellan individer som delar en CFTR -mutationsallel. Human Genetics, 98 (3), 304-309
- Svälja, m. TILL., Leaver, a. L., Christiansen, f. T., Witt, c. S., Abraham, L. J., & Dawkins, r. L. (1992). Ancestral Haplotyper: Conserva Population MHC Haplotyper. Human Immunology, 34 (4), 242-252.
- Stipendiater, m. R., Hartman, T., Hermelin, D., Landau, g. M., Rosamond, f., & Rozenberg, L. (2009, juni). Haplotypinferens begränsad av troliga haplotypdata. I årligt symposium om Combinatorial Pattern Matching (PP. 339-352). Springer, Berlin, Heidelberg.
- Gabriel, s. B., Schaffner, s. F., Nguyen, h., Moore, j. M., Roy, J., Blumenstiel, b.,... & Liu-Cordero, S. N. (2002). Strukturen för haplotypblock i det mänskliga genomet. Science, 296 (5576), 2225-2229.
- International Hapmap Consortium. (2005). En haplotypkarta över det mänskliga genomet. Nature, 437 (7063), 1299.
- Wynne, r., & Wilding, C. (2018). Mithochondrial DNA -haplotypdiversitet och ursprung för fångst sandtigerhajar (Cararias Oxen). Journal of Zoo and Aquarium Research, 6 (3), 74-78.
- Yoo och. J., Tang, j., Kaslow, r. TILL., & Zhang, K. (2007). Haplotype slutsatsen för genotypdata för nuvarande. Med hjälp av tidigare identifierade haplotyper och haplotypmönster. Bioinformatics, 23 (18), 2399-2406.
- Young, n. S. (2018). Aplastisk anemi. The New England Journal of Medicine, 379 (17), 1643-1656.
- « Hollandiska arvegenskaper, funktioner för gener, degeneration
- Lentén större egenskaper, livsmiljöer, egenskaper, vård »

